출처: BioINpro 122호
신의철/KAIST 의과학대학원 교수, 임옥재/티쎌로지(주) 대표이사
1. 환자맞춤형 암백신과 신생항원
최근 치료제의 임상연구 및 개발 분야에서 성장률과 규모 면에서 전세계적으로 가장 주목받고 있는 적응증으로 항암제, 면역치료제, 비만치료제 등을 꼽을 수 있다(그림 1).
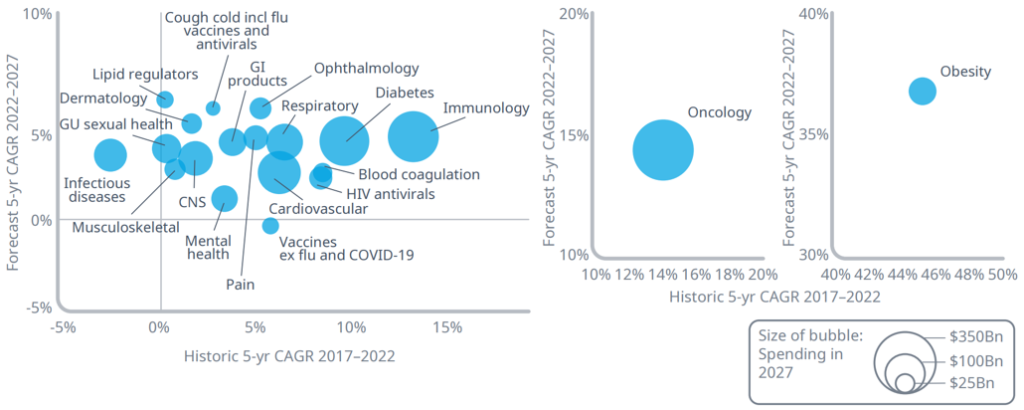
그림 1. 상위 20개 치료분야의 과거와 미래 5년 성장률 경향성
출처: IQVIA Forecast Link, IQVIA Institute, Nov 2022
그중에서 항암제 시장은 그 규모가 2022년 1960억 달러에서 2027년 3750억 달러로 급성장할 것으로 예상되는 주요 분야인데(그림 2), 그러한 항암치료제 임상의 연구 및 개발을 주도하고 있는 것이 항암 면역치료제이다. 항암 면역치료 분야에서는 PD-1/PD-L1 등의 면역관문 수용체를 타겟으로 한 면역관문 억제제를 필두로 항체-약물 접합체(antibody-drug conjugates), 이중항체(bispecific antibody) 등 항체 치료제와 CAR-T가 주도하는 세포치료제가 성장을 이끌고 있다. 하지만, 이러한 항암 면역치료제들도 아직까지는 임상에서의 치료효능에 한계가 있어 모든 암 환자들에서 충분한 효과를 나타내는 것은 아니다.
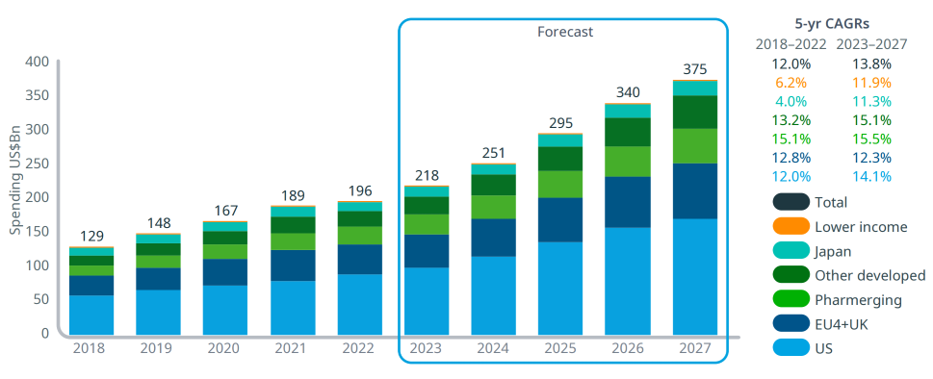
그림 2. 항암치료제 시장 예측
출처: IQVIA Oncology Link, Apr 2023
임상에서 충분하면서 지속적인 치료효과를 유도하면서 항암 면역치료제의 한계를 상호 보완하거나 극복할 수 있는 전략으로 환자맞춤형 암백신(또는 개인맞춤형 암백신, personalized cancer vaccine)에 대한 관심이 급증하고 있다. 특히 코로나19 팬데믹 와중에 큰 관심을 받게 된 mRNA 백신 기술이 향후 적용될 분야로서 환자맞춤형 암백신이 주목되면서 이를 개발하려는 바이오 산업체들도 지속적으로 늘어나고 있다. 환자맞춤형 암백신은, 각 암환자의 암세포에 존재하는 돌연변이를 파악하고 이러한 돌연변이들, 즉, mutanome 내에서 T세포 항원으로 작용할 수 있는 부분을 찾아내어 이를 백신항원으로 도입한 암 치료백신을 제조하고 환자에게 투여하는 전략이다. 이때, 돌연변이로 인해 생성된 항원 단백질 부분을 신생항원(neoantigen)이라고 하며, 신생항원은 기본적으로 MHC 분자에 결합하여 T세포를 자극하는 T세포 에피토프(neoepitope)를 포함하고 있다(그림 3).
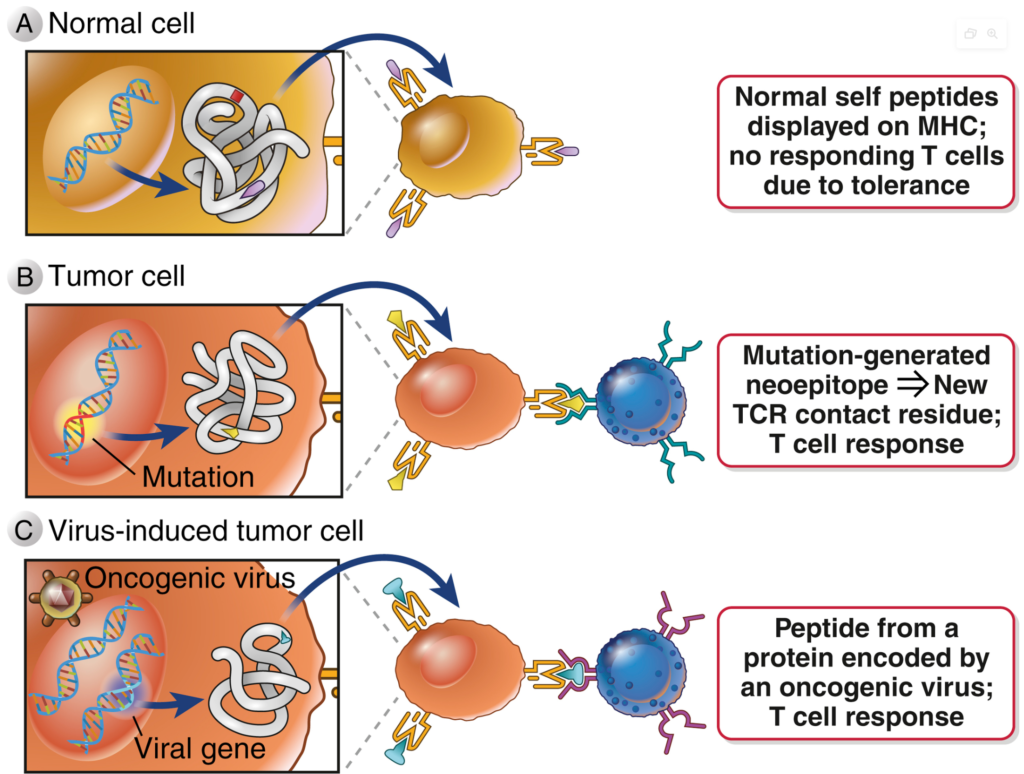
그림 3. 암세포 신생항원 및 T세포 반응
출처: Abbas, Abul, K. et al. Cellular and Molecular Immunology, 10th Edition
치료용 암백신의 경우 지난 50여 년간 다양한 임상 연구가 시도되어 왔다(Finn et al., 2018). 암항원 특이적인 CD8+ T세포 반응을 유도하거나 강화하기 위해 암특이 항원을 선별하고, 여기에 adjuvant를 도입하거나 효과적인 delivery vector를 디자인하는 등 암백신 투여 기술에 대한 다양한 임상연구가 진행되어 일부 효과를 확인하기도 했다. 그러나, 초기의 이러한 전략들은 면역저해요인들에 대한 한계를 극복하지 못해 충분한 T세포 반응을 유도하는데는 성공하지 못했다. 최근에 널리 보급된 차세대 염기서열 분석법(next generation sequencing, NGS) 덕분에 이제는 각 환자의 암 조직에 있는 유전자 돌연변이들을 매우 빠르고 정확하게 파악할 수 있는 기반이 마련됨으로써, 환자맞춤형 신생항원 발굴에 기반한 치료백신 임상연구가 보다 활기를 띠고 있다. 게다가, 선별된 항원의 백신 효능이 백신 단독 투여만으로는 충분한 항암 효력을 기대하기 어려운 경우 면역관문억제제를 동시에 투여하는 병용투여(combination therapy) 전략을 통해 그 치료 효과의 증대를 기대할 수도 있게 되었다(그림4).
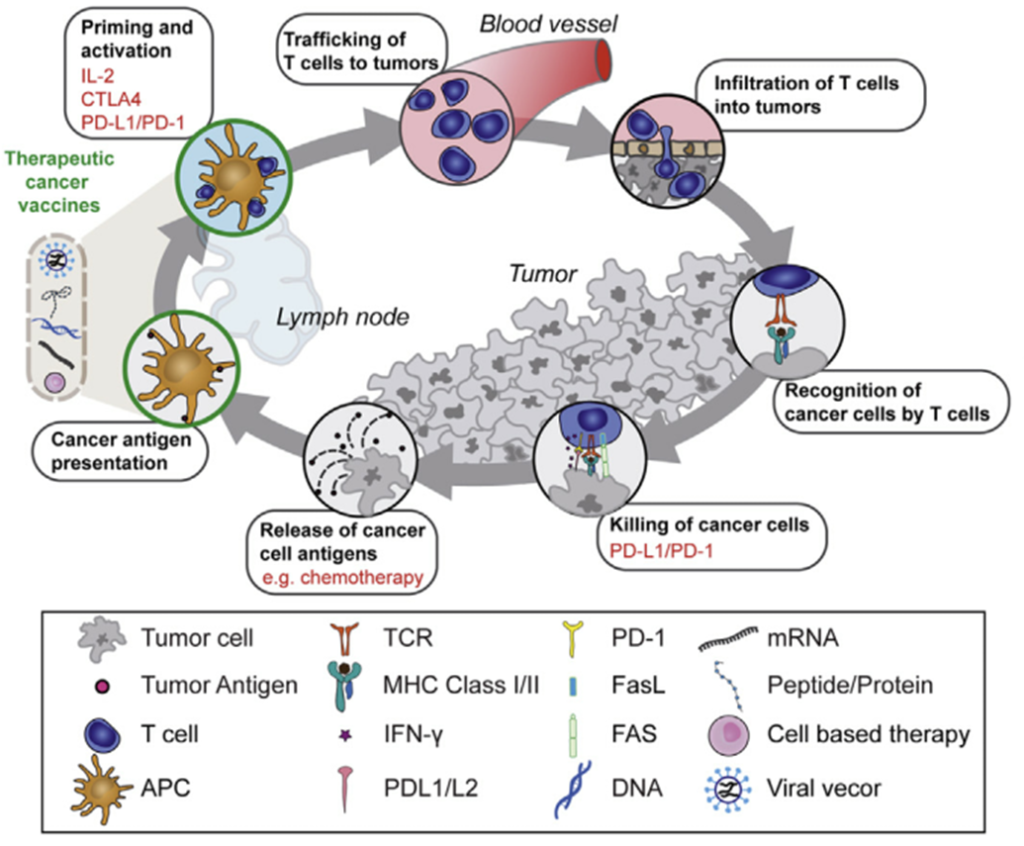
그림 4. 종양 면역에서 성공적으로 항종양 T 세포 반응 유도하기 위한 치료전략
출처: Shemesh et al. 2021 Molecular Therapy
환자맞춤형 암백신의 제작은 기본적으로 다음과 같은 3단계의 과정으로 요약해볼 수 있다(그림5).
1단계) 각 환자 암조직에서 NGS 유전자 염기서열을 분석을 하여 mutanome 정보 확보
2단계) 각 환자의 mutanome으로부터 신생항원 T세포 에피토프를 예측하기
3단계) 신생항원 T세포 에피토프를 이용하여 백신 제조하기 (이때, DNA, mRNA, 펩타이드, 수지상세포 등 다양한 백신 플랫폼 기술 활용 가능)
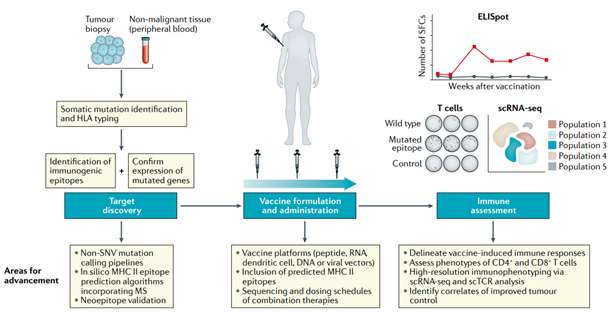
그림 5. 알고리즘 기반의 환자맞춤형 신생항원 백신 제작 과정
출처: Eryn Blass and Patrick A. Ott, 2021, Nat.Rev.Clin.Oncol.
이러한 신생항원 T세포 에피토프를 이용한 환자맞춤형 암백신의 기본적인 개념은 이미 여러 그룹에서 치료백신으로서의 가능성을 확인하고 임상연구 단계에 진입해 있다(Ott et al., 2017; Sahin et al., 2017). 신생항원을 선별하여 백신개발을 진행하고 있는 회사는 대표적으로 BioNTech, Moderna, Neon Therapeutics, Gritstone Oncology, Genocea 등을 들 수 있다. 이들 회사는 펩타이드, RNA, DNA, 면역세포 등의 다양한 플랫폼의 형태로 백신으로 개발하여 경쟁하고 있는데, 백신 단독 요법 전략을 쓰기도 하지만 Genentech, Merck, BMS 등과 공동 연구로 면역관문억제제를 병용투여하는 임상연구도 진행하고 있다. 여기서, 이들 회사의 신생항원 선별 방법을 살펴보면 공용 예측법(예, NetMHC)이나 데이터베이스(예, IEDB)를 활용하고 있는 경우가 많으며, 일부에서는 자체적으로 개발한 분석 알고리즘을 활용한 전략을 이용하는 경우도 있다(표 1).
표 1. 회사별 환자맞춤형 항암 백신 전략
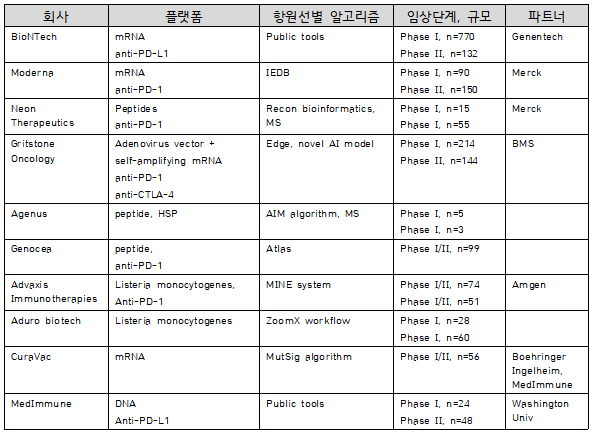
출처: Shemesh et al. (2021) Molecular Therapy
2. 신생항원 에피토프 예측기술의 현황
암치료에 있어서 환자맞춤형 암백신이 주목받고 있지만, 신생항원을 정확하게 찾아내고 검증하는 부분은 여전히 어려운 문제로 남아있다. 현재 활용할 수 있는 신생항원 에피토프 예측 알고리즘은 수많은 에피토프 후보를 제시해주지만, 이 중 아주 소수만이 환자들에서 진정한 항암 반응을 유도한다는 것이 확인되고 있다. 이는 신생항원 에피토프 예측과 검증이 일상화되고 환자맞춤형 면역요법이 현실적으로 임상에 도입되기 위해서는 더 많은 연구가 필요하다는 것을 의미한다(Nat. Biotech, 2017, 35(2):97).
앞서 언급한 것처럼 신생항원 에피토프를 선별하기 위한 첫 단계는 일반적으로 암조직과 정상조직으로부터 엑손(exon) 시퀀싱 결과를 비교해서 변이된 유전자를 구분하는 과정으로부터 시작한다. 또한, 여기에 신생항원의 발현량에 따른 가중치를 보기 위해 전사체(transcriptome) 데이터를 확인하기도 한다. 이러한 변이정보로부터 찾아내야 할 가장 중요한 부분이 T세포 반응을 유도할 수 있는 에피토프의 선별이다. 즉, 신생항원 단백질이 프로테아좀(proteasome)에 의해 펩타이드 조각으로 잘려서 TAP (ER로 펩타이드를 전달해주는 transporter)을 통해 소포체(ER)로 이동하고, ERAP (ER 내에 존재하는 amino-peptidase)에 의해 8~10개의 아미노산 잔기의 펩타이드 조각으로 추가로 잘린 후, 새롭게 합성된 MHC-I에 결합해서 세포 표면에 제시될 경우, 그러한 에피토프-MHC 복합체를 인지하는 T세포 반응을 유도할 수 있는 에피토프를 선별해야 하는 것이다(그림 6).
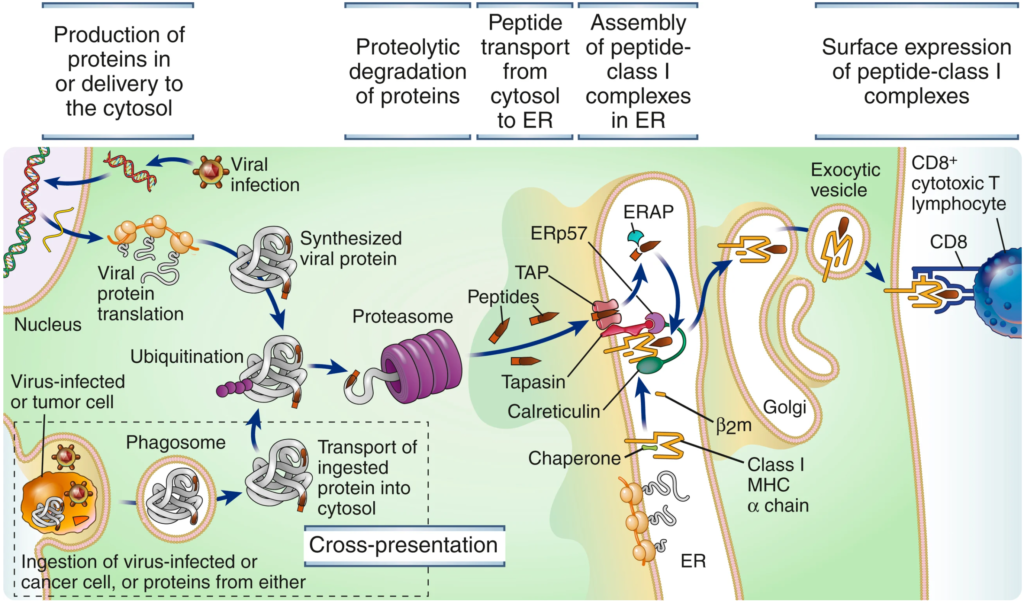
그림 6. MHC-I 항원제시 과정
출처: Abbas, Abul, K. et al. Cellular and Molecular Immunology, 10th Edition
일례로, 항원이 어떻게 에피토프 조각으로 만들어지는지에 가중치를 둔 NetChop과 같은 방식이나 세포 내에서 펩타이드 조각의 이동 조건에 가중치를 둔 NetCTL 등의 다양한 예측 방법이 알려져있기는 하나, 최근까지도 어떤 아미노산 서열의 펩타이드가 MHC-I 분자에 결합하는지를 모델링하는 데 가중치를 둔 예측방식이 임상에서 가장 널리 이용되고 있다.
MHC-I 에피토프 예측 알고리즘으로 NetMHC4.0, NetMHCpan4.0, IEDB (Immune Epitope Database) Consensus, MHCflurry 등이 가장 많이 활용되는데, 이러한 예측프로그램은 in vitro 실험 데이터 및 mass spectrometry (MS)를 통해 확보된 데이터를 통해 트레이닝되어 최대 170여 개의 HLA alleles(서로 다른 개인이 가질 수 있는 HLA 유형, HLA는 인간의 MHC를 지칭함)에 대한 에피토프 정보를 nM기반의 binding affinity로 제시해 준다.
그러나, MHC에 결합하는 펩타이드가 모두 T세포 반응을 유도하는 것은 아니라는 점을 간과해서는 안 된다. 따라서, T세포 에피토프 예측의 정확도를 향상시키기 위해서는 MHC 결합가능여부에서 나아가, 펩타이드-MHC 복합체의 T세포 수용체(TCR) 결합 여부를 예측하기 위한 다양한 고려사항들이 반영되어야 한다. 이를 위해서는 아미노산 서열 및 구조가 만드는 물리·화학적 특성뿐만 아니라, self-MHC 및 자가항원에 대한 TCR의 selection 기전과 관련된 요소도 반영될 필요가 있다. 최근까지도 개발되어 활용되고 있는 생물정보학적 접근 방식의 알고리즘을 보면 펩타이드의 특성 관련 정보 외에도, SNV (single nucleotide variations), indels 정보 등에 가중치를 두는 등 예측 정확도를 높이기 위한 노력이 계속되고 있다(표 2).
표 2. 신생항원 예측을 위한 생물정보학 방법
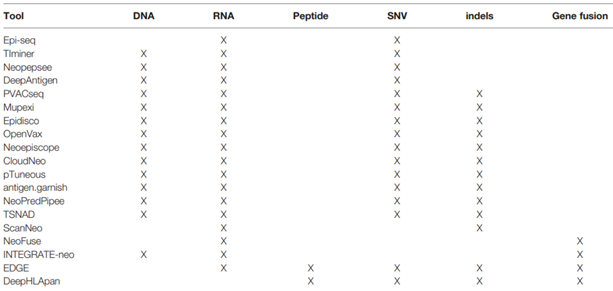
출처: Anna-Lisa Schaap-Johansen, et al. (2021) Front. Immunol.
일부에서는 에피토프의 T세포 반응을 예측하는데 있어서 사이토카인 반응에 대한 요소를 보강하기 위한 노력도 진행되고 있다(표 3). 이는 IEDB에 축적된 실험 데이터를 근거로한 머신러닝(machine learning, 기계학습) 기반의 예측 방식으로, 치료용 암백신의 디자인 전략에 각 에피토프의 IFN-γ나 IL-10 반응 예측에 대한 가중치를 부여할 수 있으나, 대부분의 경우 감염질환에 대한 백신제작 시 고려되는 사이토카인 반응을 예측하는데 활용되고 있고, 데이터의 규모나 정확도 면에서 극복할 부분이 많이 남아있다.
표 3. 사이토카인 유도 펩타이드 예측 프로그램
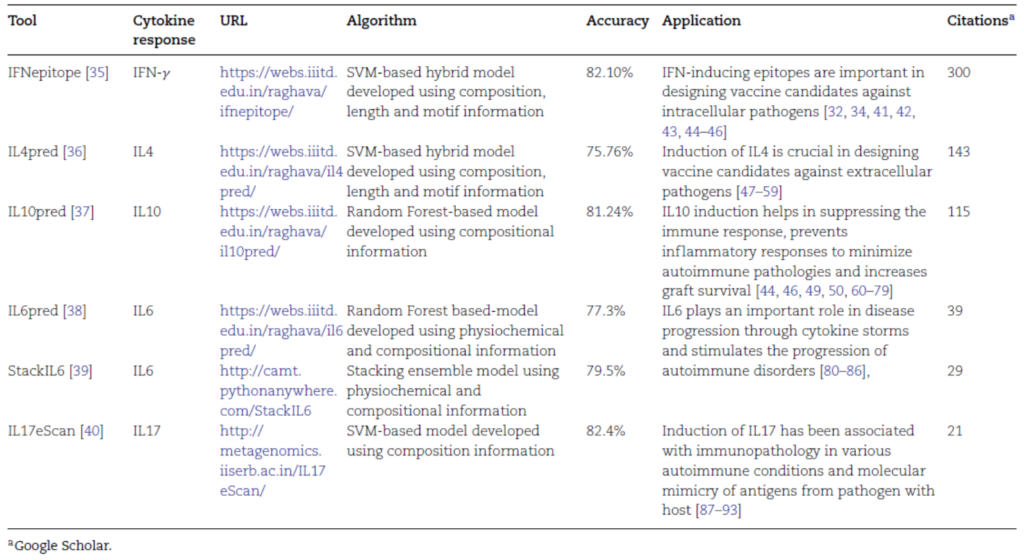
출처: Sandeep Kumar Dhanda et al. (2022) Briefings in Bioinformatics.
신생항원에서 T세포 에피토프를 예측하는데서 한 발 나아가 특정 에피토프-MHC 복합체에 결합하는 TCR을 예측하는 모델에 대한 연구도 제한적이지만 진행되고 있다(표 4). TCR 예측은 상당히 어려운데 그 이유는, 인구 집단에서 워낙 다양한 TCR이 존재하기 때문이다. 한 개인이 특정 시점에 지니고 있는 TCR의 다양성을 500만 개가량으로 추산하고, 전 세계 약 80억 인구가 11%의 TCR을 공유한다고 가정했을 때, 약 3.6 × 1015개의 TCR pool이 존재하게 된다. 이때, 자가항원이나 신생항원이 34,000종 이상의 HLA alleles에 결합하여 TCR과 반응하는 경우의 수는 포함되지 않았다(Hudson et al., 2023). 이렇게 TCR-에피토프-MHC 항원 결합 예측모델의 복합성은 현재 기술로는 풀기 어려운 난제이나, 단일세포기술 및 기계학습과 같은 디지털 생물학의 발전으로 미래에는 조금씩 실마리가 풀릴 것으로 예상된다.
표 4. TCR 에피토프 예측 프로그램(2020년 이후)
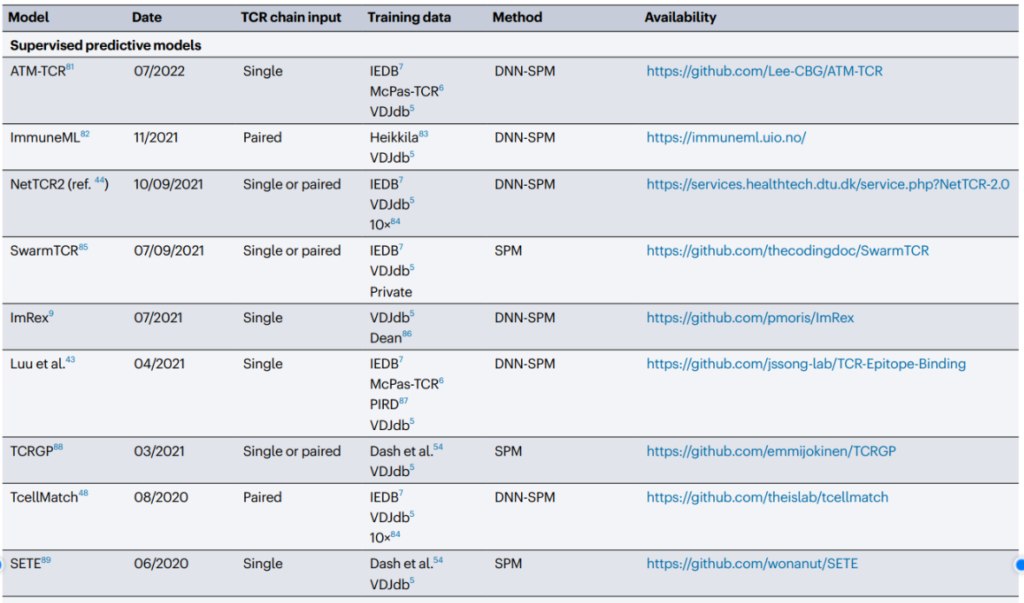
출처: Dan Hudson et al. (2023) Nature reviews Immunology
3. 신생항원 에피토프 예측기술 향상을 위한 체크 리스트
지금까지 개발된 에피토프 예측 알고리즘은 그동안 축적된 실험 데이터에 NGS 분석 데이터를 활용한 기계학습 기반 방식으로 대부분 고안되었다. 기계학습을 구현하기 위해서는 기준이 되는 독립변수(feature, 개별적이고 측정가능한 속성)를 결정하고 충분한 규모의 데이터를 확보해야 한다. 그러나, 현재 예측기술의 정확도는 그리 높지 않은 것이 현실이다. 일례로 Parker Institute가 주도하여 30여 그룹의 대학, 연구소, 회사가 Tumor Neoantigen Selection Alliance (TESLA)라는 컨소시엄을 구성하고 환자의 종양에서 DNA 변이와 관련된 신생항원 에피토프를 정확하게 예측할 수 있는 소프트웨어를 구축하기 위한 노력을 기울였다. 이 연구 결과를 2020년 10월에 Cell 학술지에 발표하였는데, 608개의 예측된 신생항원 에피토프들 중 실제로 환자의 T세포와 결합한 것은 37개로 확인되었다. 예측된 신생항원 에피토프들 중에서 불과 6%만이 T세포와 결합한 것이다(그림 7). 이렇게 낮은 효율의 신생항원 에피토프 예측결과는 효과적인 환자맞춤형 암백신의 개발에 큰 장벽이 되고 있다.
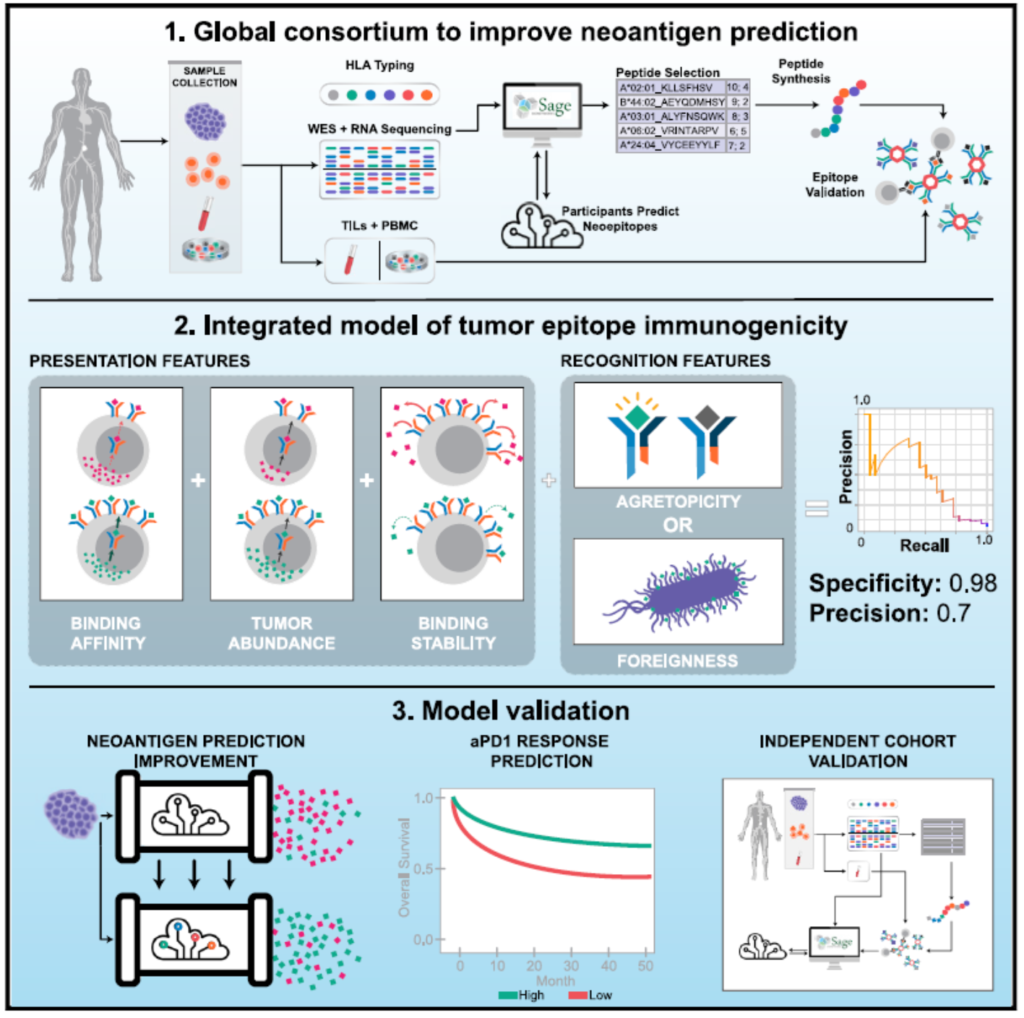
그림 7. 신생항원 예측 기술 향상을 위한 컨소시움 연구
출처: Wells et al. (2020) Cell
현재의 신생항원 에피토프 예측기술이 한계를 보이는 가장 중요한 이유는 예측 알고리즘을 만들기 위해 이용한 T세포 에피토프들의 데이터가 제한적이기 때문이다. 기계학습이라는 인공지능의 수단을 이용하여 정확도가 높은 예측 알고리즘을 만들기 위해서는 학습시킬 데이터베이스의 크기가 충분히 커야 하는데, HLA allele의 다양성에 비해서 현재 알려진 T세포 에피토프들의 데이터가 충분히 많지 않기 때문에 생기는 문제점이다. 일례로, 북미와 유럽을 포함해서 글로벌 분포도가 매우 높은 HLA-A*02:01의 경우에, 상대적으로 많은 에피토프 관련 연구 결과들이 보고되어왔고, 이러한 연구 경험을 근거로 HLA-A*02:01를 지닌 환자를 대상으로 한 임상 연구도 오랜 기간 다양한 그룹에서 시도되어 왔다. 그러나, 임상 시도가 집중된 HLA-A*02:01에 대한 연구마저도 복잡하고 정교한 에피토프의 생성(processing) 및 표지(presentation) 기전을 예측하고, 더 나아가 T세포 수용체 결합 반응까지 예측하기 위한 데이터 확보 측면에서는 성공하지 못했다. 고빈도의 HLA의 경우도 이처럼 제한적이므로 저빈도의 HLA의 경우는 에피토프 예측가능성이 매우 희박해질 수밖에 없다.
여기에 특정 HLA에 대한 에피토프가 실험 데이터로 확인이 된 경우라도 그 에피토프가 해당 HLA를 지닌 사람에서 모두 T세포 반응을 보이는 것은 아니다(그림 8). 해당 에피토프에 반응하는 T세포가 개체 내에 존재하지 않거나 검출이 어려울 만큼 활성화되지 못하는 조건에 놓여있을 가능성이 있다.
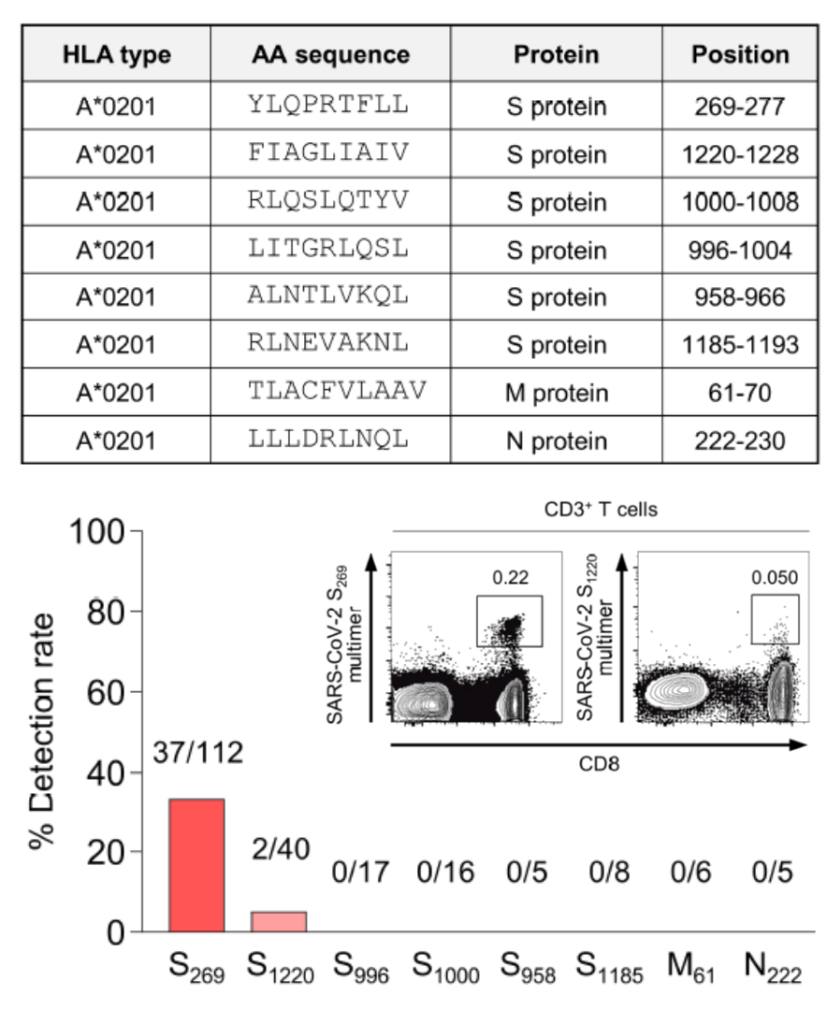
그림 8. HLA-A*02:01 multimer를 이용한 SARS-CoV-2 에피토프 결합 T세포 검출 결과
출처: Rha et al. (2021) Immunity
이렇게 제한적인 정보를 통해서 부정확하게 예측된 신생항원 에피토프를 이용하여 환자맞춤형 암백신을 제조하여 암환자에게 투여하면 암세포에 대해 작동할 T세포 반응을 유발하지 못하는 문제만 있는 것이 아니다. 부정확하게 예측된 신생항원 에피토프가 만약 MHC-I 단백질에는 결합하지만 T세포 수용체에는 결합하지 못한다면, 이러한 신생항원 에피토프는 제대로 예측된 신생항원 에피토프가 MHC-I 단백질에 결합하는 것을 경쟁적으로 방해해 오히려 암백신의 효능을 저하시킬 것이다. 즉 잘못 예측된 신생항원 에피토프를 기반으로 한 환자맞춤형 암백신은 항암 기능을 수행할 T세포를 자극하지 못하는 데에 그치지 않고 오히려 T세포 반응을 저하시킬 수도 있는 것이다(그림 9). 따라서 MHC 결합능 뿐만 아니라 T세포 반응 유도능이 있는 에피토프들을 정확하게 선별하지 않고, 예측된 다수를 모두 백신으로 제작하는 것은 오히려 백신 효능저하를 일으킬 수 있다.
그림 9. MHC-I에 경쟁적으로 결합하는 펩타이드에 의한 T세포 반응 저해 효과
출처: You et al. (2014) Immune Network 3:86-93
4. 신생항원 에피토프 예측기술의 미래
현재의 신생항원 에피토프 예측기술이 가진 한계를 정확하게 이해하고 이를 극복하기 위한 논의가 차츰 이루어지면서 신생항원 에피토프 예측기술을 개선시키기 위한 노력들이 진행되고 있다.
신생항원 에피토프 예측기술이 한계를 보이는 가장 중요한 이유로 기계학습에 필요한 데이터베이스 확보가 부족하다는 부분을 강조했다. 이를 해결하기 위해서는, T세포 에피토프들을 새롭게 발굴하고 실험적으로 검증하여 T세포 에피토프들의 데이터베이스 크기를 충분히 늘리고 예측 알고리즘을 만드는 것이 중요하다. 최근 이런 시도를 하는 연구개발이 몇몇 기업에서 이루어지고 있다.
T세포 에피토프의 데이터베이스 확충 시 반드시 고려해야 할 부분이 HLA allele의 다양성에 대한 연구이다. 앞에서 HLA-A*02:01를 예로 언급했듯이, 인구집단에 많은 HLA allele에 대해서는 제한적이나마 어느 정도 예측기술이 작동하지만, 드문 HLA allele에 대해서는 그 정확도가 현저히 떨어진다. 이는 각 HLA allele에 결합하는 T세포 에피토프들의 데이터베이스 크기의 차이로 이해될 수 있다. 즉 인구집단에 드물게 존재하는 HLA allele들에 대해서는 알려진 T세포 에피토프들이 충분히 많지 않으니 이를 기반으로 만들어진 알고리즘의 정확도는 당연히 낮을 수밖에 없는 것이다. 이 문제점도 데이터베이스의 크기를 늘림으로써 해결될 수 있다. 즉 드문 HLA allele들에 결합하는 T세포 에피토프들을 새롭게 발굴하고 실험적으로 검증하여 데이터베이스 크기를 충분히 늘리고 예측 알고리즘을 만들면 해결될 수 있을 것이다.
현재의 신생항원 에피토프 예측기술이 한계를 보이는 또 다른 이유는, 현재의 기술이 ‘어떤 8-10개 아미노산 크기의 펩타이드 조각이 MHC 단백질에 결합하기만 하면 T세포 수용체와의 결합은 무조건 일어날 것’이라는 잘못된 가정하에 개발된 것이라는 점에 있다. 하지만 실제 면역학 실험 결과를 보면, MHC 단백질에 결합한 펩타이드 조각들 중에 일부만이 T세포 수용체와 결합하여 면역반응을 유발할 수 있다. 위에서 서술한 대로, 만약 MHC 단백질에는 결합하지만 T세포 수용체에는 결합하지 못하는 펩타이드를 항원으로 사용하면 제대로 예측된 신생항원 에피토프가 MHC 단백질에 결합하는 것을 경쟁적으로 방해해 오히려 암백신의 효능을 저하시킬 것으로 예상할 수 있다. 이러한 문제점을 극복하기 위한 연구들도 수행되고 있다. 구체적으로 말하자면, T세포 에피토프 데이터베이스를 구축할 때, MHC 단백질 결합 여부만을 고려하지 않고 T세포 수용체 결합 여부까지 고려하여 더 정확한 알고리즘을 개발하고자 하는 연구개발이 진행되고 있다.
위에서, 현재의 신생항원 에피토프 예측기술이 한계를 가지는 3가지 주요한 이유를 살펴보고 이를 극복하기 위해 어떤 노력들이 진행 중인지 간단히 살펴보았다. 이러한 노력들을 통해 신생항원 에피토프 예측기술의 정확도를 획기적으로 높인다면 환자맞춤형 암백신의 효능도 획기적으로 증가할 것이다. 미래 의학의 핵심 키워드라고 할 수 있는 정밀의료(precision medicine)의 개념을 가장 이상적으로 구현한 아이디어가 바로 환자맞춤형 암백신이다. 정확한 신생항원 에피토프 예측기술의 개발과 이를 이용한 환자맞춤형 암백신 개발을 통해 암을 정복할 수 있을 것으로 기대한다.
참고문헌
The Global Use of Medicines 2023 (Outlook to 2027), IQVIA Forecast Link, IQVIA Institute Report, Jan 18, 2023
Global Oncology Trends 2023 (Outlook to 2027) IQVIA Institute Report, May 2023
Abbas, Abul, K. et al. (2021) Cellular and Molecular Immunology. 10th Edition. Elsevier.
Finn, O. (2018) The dawn of vaccines for cancer prevention. Nat Rev Immunol 18: 183–194
Eryn Blass and Patrick A. Ott, (2021) Advances in the development of personalized neoantigen-based therapeutic cancer vaccines. Nat.Rev.Clin.Oncol. 18:215-229
Ott, P., Hu, Z., Keskin, D. et al. (2017) An immunogenic personal neoantigen vaccine for patients with melanoma. Nature 547:217–221
Sahin, U., Derhovanessian, E., Miller, M. et al. (2017) Personalized RNA mutanome vaccines mobilize poly-specific therapeutic immunity against cancer. Nature 547:222–226
Nielsen M, Lundegaard C, Worning P, et al. (2003) Reliable prediction of T-cell epitopes using neural networks with novel sequence representations. Protein Sci. 12(5):1007–17.
Jurtz V, Paul S, Andreatta M, et al. (2017) NetMHCpan-4.0: improved peptide-MHC class I interaction predictions integrating eluted ligand and peptide binding affinity data. J Immunol Baltim Md. 1950(199):3360–8.
Moutaftsi M, Peters B, Pasquetto V, et al. (2006) A consensus epitope prediction approach identifies the breadth of murine T(CD8+)-cell responses to vaccinia virus. Nat Biotechnol. 24(7):817–9.
O’Donnell TJ, Rubinsteyn A, Laserson U. MHCflurry 2.0: improved pan-allele prediction of MHC class I-presented peptides by incorporating antigen processing. Cell Syst 2020;11(4):418–9.
Anna-Lisa Schaap-Johansen, et al. (2021) T cell epitope prediction and its application to immunotherapy. Front. Immunol. 12:712488
Sandeep Kumar Dhanda et al. (2022) Not all T cell epitopes are equally desired: a review of in silico tools for the prediction of cytokine-inducing potential of T-cell eptiopes. Briefings in Bioinformatics. 23(5):1-10
Dan Hudson et al. (2023) Can we predict T cell specificity with digital biology and machine learning? Nature reviews Immunology. 23:511-521
Lin, M.J., Svensson-Arvelund, J., Lubitz, G.S. et al. (2022) Cancer vaccines: the next immunotherapy frontier. Nature Cancer 3:911–926
Colby S. Shemesh, Joy C. Hsu, Iraj Hosseini, et al. (2020) Personalized cancer vaccines: clinical landscape, challenges, and opportunities. Molecular Therapy 29(2):555-570
Daniel K Wells, et al. (2020) Key Parameters of Tumor Epitope Immunogenicity Revealed Through a Consortium Approach Improve Neoantigen Prediction. Cell. 29;183(3):818-834.e13.
Yoon Seok Choi, Dong Ho Lee and Eui-Cheol Shin (2014) Relationship between Poor Immunogenicity of HLA-A2-Restricted Peptide Epitopes and Paucity of Naive CD8+ T-Cell Precursors in HLA-A2-Transgenic Mice. Immune Network 14(4):219-225
Jihye Kim, et al. (2018) Innate-like Cytotoxic Function of BystanderActivated CD8+ T Cells Is Associated with Liver Injury in Acute Hepatitis A. Immunity 48:161-173
Min-Seok Rha, et al. (2021) PD-1-Expressing SARS-CoV-2-Specific CD8+ T Cells Are Not Exhausted, but Functional in Patients with COVID-19. Immunity 54:44-52
Sooseong You, et al. (2013) Priming of Autoreactive CD8+ T Cells Is Inhibited by Immunogenic Peptides Which Are Competitive for Major Histocompatibility Complex Class I Binding. Immune Network 13(3):86-93