출처: 한국경제 바이오인사이트 매거진 (2022년 9월호)
글 신의철 (카이스트 의과학대학원 교수), 임옥재 (티쎌로지(주) 대표이사)
개인 맞춤형 암백신이 상용화되기까지는 많은 허들이 남아 있다. 그중 가장 큰 과제는 신생항원의 발굴이다. 환자 개개인이 가지고 있는 특이적인 암 돌연변이는 차세대 염기서열 분석 (NGS)를 이용하면 금방 찾을수 있지만, 이중 ‘진짜’ 면역원성을 일으키는 돌연변이는 매우 드물다. 면역원성 신생항원을 발굴하는 것이 왜 어려운지, 어떤 작업이 필요한지를 살펴봤다.
미래에 유망한 항암 면역치료의 한 가지로서 최근 개인맞춤형 암백신(personalized cancer vaccine)에 대한 관심이 급증하고 있다. 그리고 개인맞춤형 암백신을 성공적으로 개발하기 위해서는 신생항원 에피토프를 정확히 예측하는 기술이 필요하다는 점이 점차 부각되고 있다.
이번 글에서는 개인맞춤형 암백신의 개발에서 신생항원 에피토프 예측이 왜 필요한지 서술한 후, 현 예측기술의 문제점을 살펴보고 미래 발전방향에 대해 전망한다.
개인맞춤형 암백신과 신생항원
암은 기본적으로 유전자 돌연변이에 의한 질병이다. 내 몸의 정상세포에 생긴 유전자 돌연변이의 결과로 세포의 증식이 적절히 제어되지 못하면 암세포가 되는 것이다. 이러한 암세포는 면역반응의 표적이 될 수 있다. 암세포는 원래 내 세포로부터 유래되지만 유전자 돌연변이에 의해 생성된 돌연변이 단백질은 내 단백질이 아닌 남의 단백질로 면역세포에게 인식될 수 있기 때문이다.
암세포에 대해 면역반응이 유발될 수 있다는 사실 때문에 암백신에 대한 연구가 시작됐다. 다만 세균이나 바이러스에 대한 백신은 예방의 목적으로 사용하는 반면, 암백신의 경우에는 암 환자에게 치료의 목적으로 투여하는 개념으로 연구되었다. 암백신을 만들기 위해서는 적당한 항원을 선택하여 이용하여야 한다. 마치 코로나19 바이러스에 대한 백신을 만들기 위해 스파이크 단백질을 항원으로 선택한 것처럼 말이다.
그렇다면 암백신을 만들 때는 항원으로 어떤 것을 이용하여야 할까. 이론적으로 가장 좋은 것은 암세포에 생기는 유전자 돌연변이가 만드는 돌연변이 단백질을 항원으로 이용하는 것이다.
이렇게 암세포에서 생성되는 돌연변이 단백질을 신생항원 단백질(neoantigen)이라 한다. 정상세포가 암세포로 되면서 새롭게 생성되는 항원이기 때문에 이런 명칭이 붙었다.
그런데 바로 이 점에 ‘개인맞춤형’의 개념이 도입된다. 왜냐하면 암세포의 유전자 돌연변이는 각 암환자마다 모두 다르게 ‘개인화돼(personalized)’ 일어나는 것이기 때문에 동일한 종류의 암이라 하더라도 모든 환자들에게서 공통적으로 나타나는 돌연변이 단백질은 없을 것이기 때문이다.
최근 널리 보급된 NGS 덕분에 이제는 각 환자의 암 조직에 있는 유전자 돌연변이들을 매우 빠르게 파악할 수 있게 됐다.
하지만 각 암환자의 유전자 돌연변이들을 잘 찾아냈다고 해서 많은 돌연변이들 중 어느 부분이 항암 면역세포의 핵심인 T세포 반응을 유발할 신생항원 에피토프가 될지를 결정하는 것은 그리 쉬운 일이 아니다. 바로 이 때문에 신생항원 에피토프 예측기술이 필요한 것이다.
신생항원 에피토프 예측기술의 문제점
위에서 살펴본 것처럼 개인맞춤형 암백신을 만들기 위해서는 각 환자의 암 조직에서 NGS 기법을 이용하여 유전자 돌연변이들을 찾아낸 후, 이 중에서 신생항원 에피토프들을 잘 예측하고 골라내야 한다. 그렇다면 신생항원 에피토프는 어떻게 예측하고 골라낼 수 있을까.
이 질문에 대한 답을 알기 위해서는 항암 면역반응의 중추 역할을 하는 CD8 T세포의 작동 방식을 이해하여야 한다(그림 1).
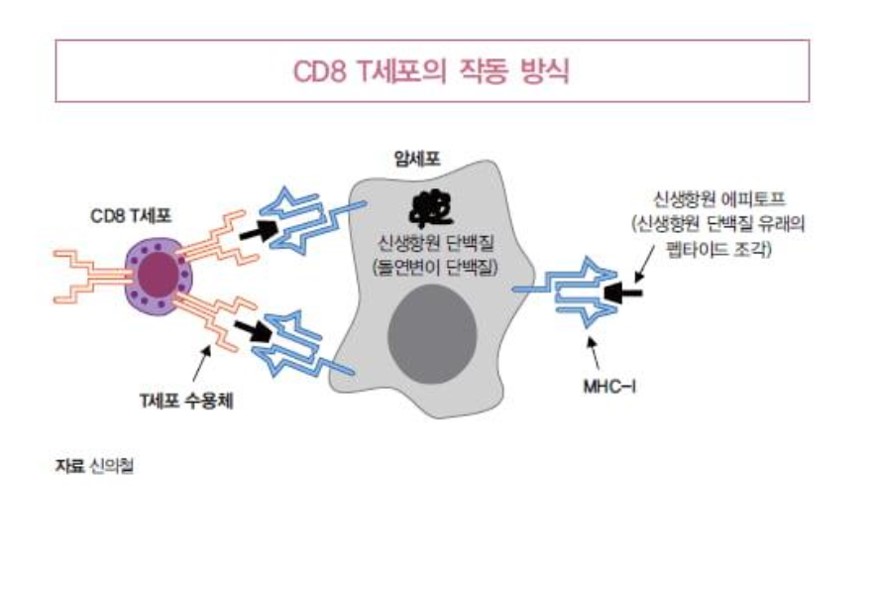
CD8 T세포는 암세포의 존재를 인지하고 이를 선택적으로 죽여서 제거하는 역할을 함으로써 항암 면역반응에 기여한다. 그런데 CD8 T세포가 암세포를 암세포라고 인지할 수 있는 것은, 암세포의 표면에 존재하는 MHC-I이라는 면역단백질 위에 결합된 신생항원 단백질 조각(8-10개의 아미노산으로 구성된 펩타이드 조각. 이를 T세포 에피토프라고 하며, 신생항원 단백질 유래의 T세포 에피토프를 신생항원 에피토프라고 함)을 CD8 T세포가 T세포 수용체를 통해 인지할 수 있기 때문이다.
이런 세밀한 기전을 고려한다면, 결국 신생항원 에피토프 예측에서 중요한 것은 암세포의 MHC-I에 결합할 수 있는 신생항원 단백질 조각을 예측하는 것이다. 즉 이번 글의 제목인 신생항원 에피토프 예측이라는 것은 ‘암세포의 돌연변이 단백질(신생항원 단백질)들 중 MHC-I에 결합할 8-10개 아미노산 크기의 펩타이드 조각(신생항원 에피토프)을 예측하고 찾아낸다는 것’을 의미한다.
신생항원 에피토프를 예측하고 찾아낸다는 것이 쉽게 보이지만 현실은 그렇지 않다. 가장 큰 문제는 MHC-I 단백질의 다양성이다. 마치 혈액형이 각 사람마다 다른 것처럼 MHC-I 단백질도 각 사람마다 다른 것을 보유하고 있다.
게다가 그 다양성은 혈액형보다 훨씬 커서 ABO 혈액형이 4가지 유형만 존재하는데 비해 MHC-I 단백질은 훨씬 더 많은 종류가 존재한다(하위 그룹 중 하나인 HLA-A의 경우만 해도 2000가지 이상의 유형이 존재한다).
이렇게 각 환자마다 MHC-I 단백질의 종류가 다르기 때문에, MHC-I 단백질에 잘 결합하는 8-10개 아미노산 크기의 펩타이드 조각의 성질도 각 환자마다 다르게 된다. 즉 각 환자마다 유전자 돌연변이가 제각기 다른데다가 MHC-I 단백질의 종류도 다르기 때문에 신생항원 에피토프를 잘 예측하고 찾아내는 것은 매우 어려운 과제인 셈이다.
신생항원 에피토프를 정확히 예측하고 찾아내는 것이 쉬운 일은 아니지만, 현재에도 신생항원 에피토프를 예측하는 기술이 존재한다. 구체적으로 말하자면, 특정 단백질의 아미노산 서열을 입력하면 특정 MHC-I 단백질에 결합할 가능성이 높은 8-10개 아미노산 크기의 펩타이드 조각을 예측해 주는 알고리듬이라고 할 수 있다.
이러한 알고리듬은 축적된 데이터를 기반으로 만들어진 것이다. 즉, 그동안의 면역학 연구를 통하여 각각의 MHC-I 단백질에 잘 결합하는 것으로 알려진 8-10개 아미노산 크기의 펩타이드 조각, 즉 T세포 에피토프들의 데이터베이스를 기반으로 하여 만들어진 알고리듬으로서, 어떤 단백질의 아미노산 서열이 주어졌을 때 어떤 펩타이드 조각이 MHC-I에 잘 결합할지 예측해 주는 것이다.
하지만 현재 기술의 정확도는 그리 높지 않다. 일례로 2020년 10월에 Cell 학술지에 발표된 Tumor Neoantigen Selection Alliance (TESLA)의 신생항원 에피토프 예측기술 결과를 보면, 608개의 예측된 신생항원 에피토프들 중 실제로 환자의 T세포와 결합한 것은 37개였다. 예측된 것들 중 불과 6%만이 T세포와 결합한 것이다.
이렇게 낮은 효율의 신생항원 에피토프 예측결과는 효과적인 개인맞춤형 암백신의 개발에 큰 장벽이 된다.
부정확하게 예측된 신생항원 에피토프 정보를 이용하여 개인맞춤형 암백신을 제조하여 암환자에게 투여하면 암세포에 대해 작동할 T세포 반응을 유발하지 못하는 데만 문제가 있는 것이 아니다. 부정확하게 예측된 신생항원 에피토프가 만약 MHC-I 단백질에는 결합하지만 T세포 수용체에는 결합하지 못한다면, 이러한 신생항원 에피토프는 제대로 예측된 신생항원 에피토프가 MHC-I 단백질에 결합하는 것을 경쟁적으로 방해해 오히려 암백신의 효능을 저하시킬 것이다. 즉 잘못 예측된 신생항원 에피토프를 기반으로 한 개인맞춤형 암백신은 항암 기능을 수행할 T세포를 자극하지 못하는 데에 그치지 않고 오히려 T세포 반응을 저하시킬 수도 있는 것이다.
신생항원 에피토프 예측기술의 미래
위에서 살펴본 대로, 현재의 신생항원 에피토프 예측기술은 아직 제한점이 많고 이를 개선시키지 않는다면 개인맞춤형 암백신의 미래도 어두울 수 밖에 없을 것이다. 하지만 다행히도 ‘현재의 신생항원 에피토프 예측기술이 왜 한계를 가지는가?’에 대한 논의가 최근 이루어지면서 신생항원 에피토프 예측기술을 개선시키기 위한 노력들이 진행되고 있다.
현재의 신생항원 에피토프 예측기술이 한계를 보이는 가장 중요한 이유는 예측 알고리듬을 만들기 위해 이용한 T세포 에피토프들의 데이터가 제한적이기 때문이다. 기계학습이라는 인공지능의 수단을 이용하여 좋은 예측 알고리듬을 만들기 위해서는 학습시킬 데이터베이스의 크기가 충분히 커야 하는데, 현재 알려진 T세포 에피토프들의 데이터가 충분히 많지 않기 때문에 생기는 문제점이다.
이를 해결하기 위해서는, T세포 에피토프들을 새롭게 발굴하고 실험적으로 검증하여 T세포 에피토프들의 데이터베이스 크기를 충분히 늘리고 예측 알고리듬을 만드는 것이 중요하다. 최근 이런 시도를 하는 연구개발이 몇몇 기업에서 이루어지고 있다.
현재의 신생항원 에피토프 예측기술이 한계를 보이는 또 다른 이유는, 현재의 기술이 ‘어떤 8-10개 아미노산 크기의 펩타이드 조각이 MHC-I 단백질에 결합하기만 하면 T세포 수용체와의 결합은 무조건 일어날 것’이라는 잘못된 가정 하에 개발된 것이라는 점에 있다.
하지만 실제 면역학 실험 결과를 보면, MHC-I 단백질에 결합한 펩타이드 조각들 중에 일부만이 T세포 수용체와 결합하여 면역반응을 유발할 수 있다.
앞서 서술한대로, 만약 MHC-I 단백질에는 결합하지만 T세포 수용체에는 결합하지 못하는 펩타이드를 항원으로 사용하면 제대로 예측된 신생항원 에피토프가 MHC-I 단백질에 결합하는 것을 경쟁적으로 방해해 오히려 암백신의 효능을 저하시킬 것으로 예상할 수 있다.
이러한 문제점을 극복하기 위한 연구들도 수행되고 있다. 구체적으로 말하자면, T세포 에피토프 데이터베이스를 구축할 때, MHC-I 단백질 결합 여부만을 고려하지 않고 T세포 수용체 결합 여부까지 고려하여 더 정확한 알고리듬을 개발하고자 하는 연구개발이 진행되고 있는 것이다.
현재 신생항원 에피토프 예측기술의 또 다른 문제점은, 인구집단에 많은 MHC-I 단백질 유형에 대해서는 어느 정도 작동하지만 드문 MHC-I 단백질 유형에 대해서는 그 정확도가 현저히 떨어진다는 점이다.
이는 각 MHC-I 단백질 유형에 결합하는 T세포 에피토프들의 데이터베이스 크기의 차이로 이해될 수 있다. 즉 인구집단에 드물게 존재하는 MHC-I 단백질 유형에 대해서는 알려진 T세포 에피토프들이 그리 많지 않으니 이를 기반으로 만들어진 알고리듬의 정확도는 당연히 낮을 수밖에 없는 것이다.
이 문제점도 데이터베이스의 크기를 늘림으로써 해결될 수 있다. 즉 드문 MHC-I 단백질 유형에 결합하는 T세포 에피토프들을 새롭게 발굴하고 실험적으로 검증하여 데이터베이스 크기를 충분히 늘리고 예측 알고리듬을 만들면 해결될 수 있을 것이다.
미래 의학의 핵심 키워드라고 할 수 있는 정밀의료(precision medicine)의 개념을 가장 멋지게 구현한 아이디어가 바로 개인맞춤형 암백신이다. 정확한 신생항원 에피토프 예측기술의 개발과 이를 이용한 개인맞춤형 암백신 개발을 통해 암을 정복할 수 있는 미래를 꿈꿔 본다.